Le LRSV héberge le plateau MetaToul-AgromiX. Ce plateau est dédié à l’analyse des métabolites végétaux ainsi qu’aux métabolites fongiques et bactériens pouvant être impliqués dans les interactions biotiques.
Le plateau MetaToul-AgromiX fait partie de la plateforme de Metabolomique et Fluxomique de Toulouse (MetaToul), plateforme technologique de la GénoToul. Elle regroupe des compétences scientifiques et techniques (chercheurs, ingénieurs, techniciens) et des moyens (équipements de pointe : Résonance Magnétique Nucléaire, Spectrométrie de Masse, Robots de préparations d’échantillons) dans le domaine de l’analyse globale du métabolisme. MetaToul met à disposition de la communauté scientifique les concepts, outils et méthodes liés à l’analyse du métabolisme dans des cellules, tissus, et organismes.
La plateforme MetaToul, labellisée IBISA, est engagée dans une démarche qualité qui constitue un outil essentiel pour l’amélioration continue de son fonctionnement et pour la qualité des services de la plateforme. Elle est certifiée ISO 9001 et NFX-50-900.
MetaToul est partenaire du projet d’Infrastructure Nationale en Métabolomique et Fluxomique MetaboHUB dirigé par Fabien Jourdan, avec les plateformes de Bordeaux, Clermont-Ferrand, Grand-Ouest et Ile de France.
Liste des services et analyses proposés par le plateau MetaToul-AgromiX
La plateforme MetaToul-Agromix peut être sollicitée par la communauté scientifique publique et privée pour des analyses qualitatives et quantitatives de métabolites végétaux et microbiens pour :
- Réaliser des prestations de service pour répondre à des besoins ponctuels
- Développer de nouvelles méthodes. Une collaboration ou un partenariat peut être élaboré dans le cadre du montage et de la mise en œuvre de projets de recherche.
- Mettre à disposition des instruments analytiques.
Hormonomics
Analyse par spectrométrie de masse de phytohormones :
| Acide Abscissique | Acide Abscissique (ABA) Ester de glucose de l’ABA (ABA-GE) … |
| Jasmonates |
Acide jasmonique (JA) Acide Jasmonique-Phenylalanine (Ja-Phe : detection uniquement), |
| Auxines | 1H-indole-3-acetic acid (AIA) Indole-3-butyric acid (IBA) 3-indolepropionic acid (IPA) N-(3-indolyacetyl)-alanine (AIA-Ala) N-(3-indolyacetyl)-aspartate (AIA-Asp) N-(3-indolyacetyl)-glycine (AIA-Gly) N-(3-indolyacetyl)-glutamate (AIA-Glu) indole-3-acétaldéhyde (IAAI) indole-3-acétamide (IAM) indole-3-acétonitrile (IAN) indole-3-pyruvate (IPyA) |
| Cytokinines |
cis et trans zéatine (cZ et tZ)
Dihydrozeatin-9-glucoside (DHZ9G) |
| Gibbérellines | GA1, GA3, GA4, GA7, GA5, GA24 … |
| Salicylates |
Acide salicylique libre (SA) |
| Strigolactones | strigol, sorgolactone, orobanchol, 5-desoxy strigol … GR24* |
| Brassinostéroïdes | brassinolide (BR) |
* Phytohormones de synthèse
Matériel et matrices biologiques pouvant être analysés :
Plantules, racines, feuilles, exsudats et milieux de culture de micro-organismes.
Medicago truncatula, Arabidopsis thaliana, …
Le protocole d’extraction est développé en fonction du matériel biologique.
Mise à disposition d’appareils et formation des utilisateurs
Le personnel du plateau peut réaliser des formations adaptées à vos besoins : préparation d’échantillons, utilisation d’un équipement analytique, retraitement de données.
Équipements du plateau MetaToul-AgromiX :
- U-HPLC-MS Q-Exactive Plus (Thermoscientific)
- LC-MS Qtrap 5500 (AB Sciex)
- GC-MS TSQ Quantum (Thermoscientific)
Activités de Recherche et Développement
Métabolomique Globale : 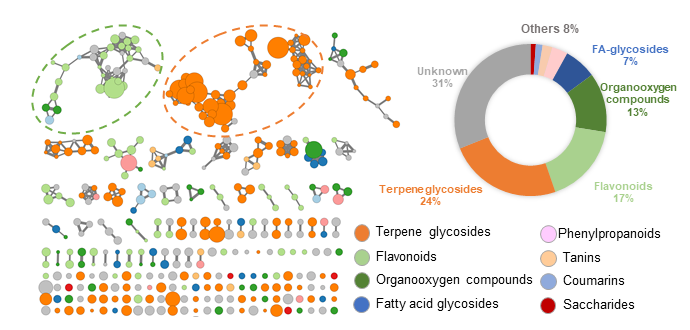
Déréplication d’un extrait complexe
Analyses multivariées (classification, régressions, analyses discriminantes)
Approches multi-omiques
Métabolomique Ciblée :
Métabolites végétaux : phytoalexines, d’alcaloïdes, composés phénoliques, flavonoïdes, …
Métabolites microbiens : lipochitooligosaccharides (LCOs), chitooligosaccharides (COS) …
Une collaboration ou un partenariat peuvent être élaborés dans le cadre de la mise en œuvre de projets de recherche.
Publications
Pedro G Vásquez-Ocmín, Guillaume Marti, Alice Gadea, Guillaume Cabanac, Juan A Vásquez-Briones, Sandro Casavilca-Zambrano, Nadia Ponts, Patricia Jargeat, Mohamed Haddad, Stéphane Bertani. 2023. ‘Metabotyping of Andean pseudocereals and characterization of emerging mycotoxins’. https://doi.org/10.1016/j.foodchem.2022.135134
Bram Danneels, Monique Blignaut, Guillaume Marti, Simon Sieber, Peter Vandamme, Marion Meyer, Aurélien Carlier. 2023. ‘Cyclitol metabolism is a central feature of Burkholderia leaf symbionts’. https://doi.org/10.1111/1462-2920.16292
Ximena Chirinos, Shiyu Ying, Maria Aurineide Rodrigues, Elie Maza, Anis Djari, Guojian Hu, Mingchun Liu, Eduardo Purgatto, Sylvie Fournier, Farid Regad, Mondher Bouzayen, Julien Pirrello. 2023. ‘Transition to ripening in tomato requires hormone-controlled genetic reprogramming initiated in gel tissue’. https://doi.org/10.1093/plphys/kiac464
Ana, Romeo-Oliván, Justine Chervin, Coralie Breton, Thierry Lagravère, Jean Daydé, Bernard Dumas, Alban Jacques. 2022. ‘Comparative Transcriptomics Suggests Early Modifications by Vintec® in Grapevine Trunk of Hormonal Signaling and Secondary Metabolism Biosynthesis in Response to Phaeomoniella chlamydospora and Phaeoacremonium minimum’. https://doi.org/10.3389/fmicb.2022.898356
Houssein, Zhour, Fabrice Bray , Israa Dandache, Guillaume Marti, Stéphanie Flament, Amélie Perez, Maëlle Lis, Llorenç Cabrera-Bosquet, Thibaut Perez, Cécile Fizames, Ezekiel Baudoin, Ikram Madani, Loubna El Zein, Anne Aliénor Véry, Christian Rolando, Hervé Sentenac, Ali Chokr, Jean-Benoît Peltier. 2022. ‘Wild Wheat Rhizosphere-Associated Plant Growth-Promoting Bacteria Exudates: Effect on Root Development in Modern Wheat and Composition’. https://doi.org/10.3390/ijms232315248
Fabio, Espichán , Rosario Rojas , Fredy Quispe, Guillaume Cabanac, Guillaume Marti. 2022. ‘Metabolomic characterization of 5 native Peruvian chili peppers (Capsicum spp.) as a tool for species discrimination’. https://doi.org/10.1016/j.foodchem.2022.132704
Chervin, Justine, Ana Romeo-Oliván, Sylvie Fournier, Virginie Puech-Pages, Bernard Dumas, Alban Jacques, and Guillaume Marti. 2022. ‘Modification of Early Response of Vitis Vinifera to Pathogens Relating to Esca Disease and Biocontrol Agent Vintec® Revealed By Untargeted Metabolomics on Woody Tissues’. Frontiers in Microbiology 13. https://www.frontiersin.org/article/10.3389/fmicb.2022.835463.
Vásquez-Ocmín, Pedro G., Guillaume Marti, Maxime Bonhomme, Fabienne Mathis, Sylvie Fournier, Stéphane Bertani, and Alexandre Maciuk. ‘Cannabinoids vs. Whole Metabolome: Relevance of Cannabinomics in Analyzing Cannabis Varieties’, 2021. https://doi.org/10.1101/2021.06.07.447363.
Vásquez-Ocmín, Pedro G., Alice Gadea, Sandrine Cojean, Guillaume Marti, Sébastien Pomel, Anne-Cécile Van Baelen, Liliana Ruiz-Vásquez, et al. « Metabolomic Approach of the Antiprotozoal Activity of Medicinal Piper Species Used in Peruvian Amazon ». Journal of Ethnopharmacology 264, 2021, 113262. https://doi.org/10.1016/j.jep.2020.113262.
Vial, Thomas, Guillaume Marti, Dorothée Missé, et Julien Pompon. « Lipid Interactions Between Flaviviruses and Mosquito Vectors ». Frontiers in Physiology 12, 2021, 763195. https://doi.org/10.3389/fphys.2021.763195.
M. Khatib, C. Pouzet, C. Lafitte, J. Chervin, V. Bonzon-Ponnet, A. Jauneau, M.-T. Esquerré-Tugayé, Phenolic profile of a Parma violet unveiled by chemical and fluorescence imaging, AoB PLANTS, 2021;, plab041, https://doi.org/10.1093/aobpla/plab041
Fraisier-Vannier, O.; Chervin, J.; Cabanac, G.; Puech, V.; Fournier, S.; Durand, V.; Amiel, A.; André, O.; Benamar, O. A.; Dumas, B.; Tsugawa, H.; Marti, G. MS-CleanR: A Feature-Filtering Workflow for Untargeted LC–MS Based Metabolomics. Anal. Chem. 2020, acs.analchem.0c01594. https://doi.org/10.1021/acs.analchem.0c01594.
Vial, T.; Tan, W.-L.; Deharo, E.; Missé, D.; Marti, G.; Pompon, J. Mosquito Metabolomics Reveal That Dengue Virus Replication Requires Phospholipid Reconfiguration via the Remodeling Cycle. Proc Natl Acad Sci USA 2020, 202015095. https://doi.org/10.1073/pnas.2015095117.
Pons, S., Fournier, S., Chervin, C., Becard, G., Rochange, S., Frei-dit-frey, N., and Puech-pages, V. Phytohormone production by the arbuscular mycorrhizal fungus Rhizophagus irregularis. PLoS ONE, 15(10):e0240886. 2020. https://doi.org/10.1371/journal.pone.0240886.
Rush, T.A., Puech-Pagès, V., Bascaules, A., Jargeat, P., Maillet, F., Haouy, A., et al. Lipo-chitooligosaccharides as regulatory signals of fungal growth and development. Nat Commun 11. 2020. http://dx.doi.org/10.1038/s41467-020-17615-5.
Bonhomme, M., Bensmihen, S., André, O., Amblard, E., Garcia, M., Maillet, F., Puech-Pagès, V. et al. Distinct genetic bases for plant root responses to molecules from distinct microbial origins. bioRxiv 33: 285668. 2020. https://doi.org/10.1101/2020.09.09.285668.
Cope, K.R., Bascaules, A., Irving, T.B., Venkateshwaran, M., Maeda, J., Garcia, K., et al. The Ectomycorrhizal Fungus Laccaria bicolor Produces Lipochitooligosaccharides and Uses the Common Symbiosis Pathway to Colonize Populus Roots. Plant Cell 31: 2386–2410. 2019. https://doi.org/10.1105/tpc.18.00676.
Volpe, V., Carotenuto, G., Berzero, C., Cagnina, L., Puech-Pagès, V., and Genre, A. Short chain chito-oligosaccharides promote arbuscular mycorrhizal colonization in Medicago truncatula. Carbohydr Polym 229: 115505. 2019. https://doi.org/10.1016/j.carbpol.2019.115505
Gibelin-Viala, C., Amblard, E., Puech-Pagès, V., Bonhomme, M., Garcia, M., Bascaules-Bedin, A., et al. The Medicago truncatula LysM receptor-like kinase LYK9 plays a dual role in immunity and the arbuscular mycorrhizal symbiosis. New Phytol 223: 1516–1529, 2019. https://doi.org/10.1111/nph.15891.
Pellegrin, C., Daguerre, Y., Ruytinx, J., Guinet, F., Kemppainen, M., Frey, N.F. dit, Puech-Pagès, V., et al. Laccaria bicolor MiSSP8 is a small-secreted protein decisive for the establishment of the ectomycorrhizal symbiosis. Environ Microbiol 21: 3765–3779. 2019. https://doi.org/10.1111/1462-2920.14727

Modalités d’accès pour une demande de projet
Un mode d’accès a été défini par la plateforme MetaToul selon le schéma suivant :
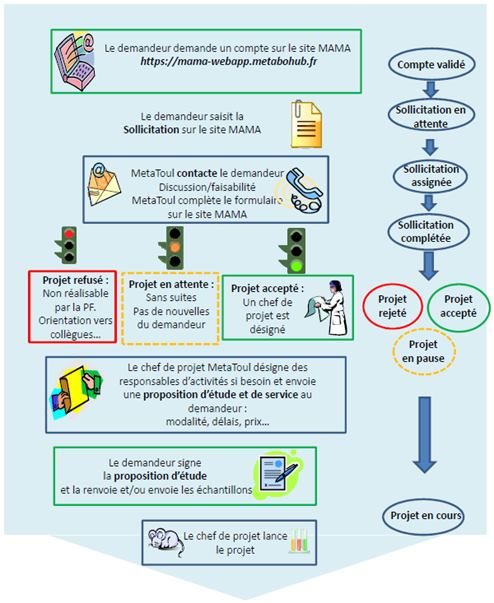
- Le demandeur prend contact avec la plateforme MetaToul en utilisant le site de demande de projet de MetaboHub (MAMA):
https://mama-webapp.metabohub.fr/
- Le responsable du plateau vous contactera pour discuter du projet afin de l’évaluer, l’accepter ou le refuser.
Le projet pourra éventuellement être renvoyé vers un autre plateau de la plateforme MetaToul ou une autre plateforme de l’infrastructure MetaboHUB.
Lorsque le projet est accepté, un chef de projet est désigné.
- Une « proposition d’étude et de service » vous sera adressée par le chef de projet. Elle contient les conditions et engagements, les modalités de réalisation, les délais et le prix.
4. Le demandeur renvoie ensuite la proposition signée au chef de projet pour commencer le projet.
Personnels
Guillaume Marti (guillaume.marti@univ-tlse3.fr) : Directeur scientifique du plateau Metatoul-Agromix (Maître de conférences en pharmacognosie, métabolomique)
Sylvie Fournier (sylvie.fournier1@univ-tlse3.fr) : Responsable technique et opérationnelle du plateau MetaToul-Agromix, Animatrice Qualité du plateau (ingénieure d’études en spectrométrie de masse)
Amélie Pérez (amélie.perez@univ-tlse3.fr) : Assitante-Ingénieure en analyse chimique
Virginie Puech-Pagès (virginie.puech-pages@univ-tlse3.fr) : Maître de conférences en biochimie, spectrométrie de masse
Recrutés sur projets :
Axel Dablanc : Ingénieur d’études bioinformatique
Solweig Hennechart : Doctorante bioinformatique
N’hésitez pas à venir nous rendre visite, à nous téléphoner ou nous envoyer un e-mail pour nous parler de votre projet !
Contact
|
contact-agromix@groupes.renater.fr Tel : +33 5 34 32 38 31 |
lrsv.gestion@univ-tlse3.fr
Phone
Standard : 05.34.32.38.01
Fax : 05.34.32.38.02
Find us
24, chemin de Borde-Rouge.
31320, Auzeville-Tolosane. FRANCE





