Notre Equipe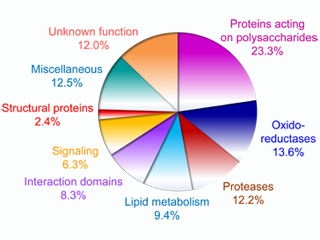
Vincent BURLAT
Professeur des Universités - Université Paul Sabatier-Toulouse 3
burlat@lrsv.ups-tlse.fr
Hervé CANUT
CR CNRS
canut@lrsv.ups-tlse.fr

Josiane CHOURRE
Adjointe technique - Université Paul Sabatier-Toulouse 3
chourre@lrsv.ups-tlse.fr
Laurent HOFFMANN
Maître de Conférences - Université Paul Sabatier-Toulouse 3
hoffmann@lrsv.ups-tlse.fr
David ROUJOL
Ingénieur d'études - Université Paul Sabatier-Toulouse 3
roujol@lrsv.ups-tlse.fr
Bastien DAUPHIN
Doctorant - Université Paul Sabatier-Toulouse 3
bastien.dauphin@lrsv.ups-tlse.fr
Hasan KOLKAS
Doctorant - Université Paul Sabatier-Toulouse 3
Sébastien VIUDES
Doctorant - Université Paul Sabatier-Toulouse 3
sebastien.viudes@lrsv.ups-tlse.fr
Anciens membres
Cécile ALBENNE (maître de conférence UPS, 2005-2016)
Kevin BELLANDE (doctorant, 2015-2018)
Edith FRANCOZ (doctorante/ATER, 2012-2016)
May HIJAZI (doctorante, 2008-2011)
Muhammad IRSHAD (doctorant, 2005-2008)
Adélaïde JACQ (doctorante, 2013-2016)
Huan NGUYEN-KIM (doctorant, 2011-2015)
Jonathas PEREIRA DAS GRACAS (doctorant Université de Sao Paulo, Brésil, 2017)
Rafael PONT-LEZICA (professeur émérite UPS)
Thèmes de Recherche
La paroi cellulaire est une structure dynamique jouant un rôle important dans la croissance, la différenciation, et les interactions avec les pathogènes ou les facteurs abiotiques de l’environnement. La paroi est à la fois source et site de perception de signaux. Nos travaux ont pour objectif de contribuer à la compréhension du rôle des protéines pariétales dans les processus de développement. Ceci nécessite (i) l’identification de l’ensemble des protéines et des gènes correspondants, (ii) le suivi de la dynamique du protéome dans différentes conditions physiologiques, et (iii) la mise en oeuvre d’analyses fonctionnelles.
Quatre thèmes de recherche sont actuellement développés :
- Protéomique
- Interactions protéines/polysaccharides
- Récepteurs lectine-kinases
- Rôle des peroxydases de classe III
Protéomique de la paroi cellulaire végétale
Contact : Elisabeth JAMET
Nous recrutons un ingénieur d’étude sur un contrat de 24 mois pour participer à notre nouveau projet de recherche financé par l’ANR, STRESS-PEPT, et dédié à une étude de peptidomique dans le cadre d’interactions plantes/microorganismes : Recrutement IE_AI_ANR Stress-Pept_07.2021
Longtemps décrite comme une enveloppe rigide autour des cellules végétales, la paroi cellulaire est maintenant considérée comme un compartiment dynamique impliqué dans de nombreux processus physiologiques tels que le développement, la signalisation ou la défense contre des stress biotiques ou abiotiques. Elle est constituée majoritairement de réseaux de polysaccharides (90% en masse) mais renferme aussi des protéines, formant un assemblage supramoléculaire complexe. Sa composition biochimique et l’agencement de ses constituants polysaccharidiques et protéiques varient tout au long de la vie de la cellule. Les protéines jouent des rôles clés dans le remaniement et la structuration de l’architecture pariétale, en modifiant les polysaccharides, générant des signaux ou en interagissant avec d’autres constituants de la paroi. Cependant, les connaissances actuelles sur les fonctions des protéines pariétales sont très incomplètes et l’enjeu des années à venir sera de décrypter plus finement leurs fonctions et de comprendre leurs interactions in muro.
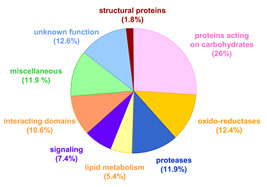
Le protéome pariétal d’A. thaliana
Depuis le début des années 2000, l’équipe s’est illustrée par ses travaux en protéomique pariétale et tient maintenant une place de leader dans ce domaine. Elle a fortement contribué à l’analyse du protéome de paroi de la plante modèle Arabidopsis thaliana qui constitue le plus grand protéome pariétal décrit à ce jour. Différents matériels ont été analysés par l’équipe : suspensions cellulaires (Borderies et al. 2003), racines (Nguyen-Kim et al. 2016), rosettes (Boudart et al. 2005, Hervé et al. 2016), tiges (Duruflé et al. 2017), secrétome de plantules étiolées en milieu liquide (Charmont et al. 2005) ou encore hypocotyles de plantules étiolées (Feiz et al. 2006, Irshad et al. 2008). Des études de glycoprotéomique ont été réalisées sur ce dernier modèle, révélant l’importance des N-glycoprotéines dans les parois (Zhang et al. 2011). L’ensemble de ces données, complétées par des données de la littérature, porte à 935 le nombre de protéines pariétales d’A. thaliana identifiées à ce jour, soit environ la moitié de ce qui est attendu (Albenne et al. 2013, Nguyen-Kim et al. 2016, Hervé et al. 2016).
Les activités de protéomique se sont étendues à d’autres plantes, telles que Brachypodium distachyon, Saccharum officinarum et triticum aestivum Le protéome pariétal de B. distachyon est à ce jour le mieux décrit parmi les monocotylédones avec 594 protéines identifiées (Douché et al. 2013, Francin-Allami et al. 2015, Francin-Allami et al. 2016). Les données de protéomique pariétale, obtenues par l’équipe ou par d’autres groupes, sont référencées dans la banque de données WallProtDB qui renferme à ce jour 3401 protéines issues de 12 espèces végétales différentes (San Clemente et Jamet 2015). Cette base de données permet d’uniformiser les annotations et la classification des protéines en neuf groupes fonctionnels. Elle est référencée parmi les OMICS tools et à Database Commons.
L’équipe a créé un outil bioinformatique complémentaire pour faciliter les analyses de protéomique. Il s’agit de ProtAnnDB qui permet de centraliser les données d’annotation structurale (prédiction de localisation sub-cellulaire) et fonctionnelle (prédiction de domaines fonctionnels) obtenues grâce à différents logiciels bioinformatiques disponibles en ligne (San Clemente et al. 2009). Cet outil permet d’annoter très rapidement les protéines identifiées grâce à la spectrométrie de masse. Il concerne actuellement 18 espèces végétales.
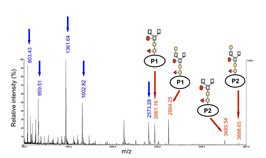
Identification de N-glycopeptides de la peroxydase At3g32980 par MALDI-TOF MS
Nos travaux ont également permis la mise en évidence de modifications post-traductionnelles de protéines pariétales en conjuguant différentes approches chromatographiques et l’exploitation des données de spectrométrie de masse (Canut et al. 2016). Nous avons ainsi localisé des acides aminés modifiés non encore décrits, des N– et des O-glycosylations et des sites de maturation de protéines (Albenne et al. 2009, Zhang et al. 2011, Hijazi et al. 2012).
Une recherche systématique de résidus hydroxyproline (Hyp) dans les données de spectrométrie de masse a permis de recenser de nouvelles familles de protéines pariétales affectées par cette modification post-traductionnelle. Un nouveau code d’hydroxylation des résidus proline (Pro) a été proposé (Canut et al. 2016, Duruflé et al. 2017). Il permet de prédire la position des résidus Hyp dans des familles de protéines riches en résidus Pro (HRGP), mais se révèle inefficace pour les autres familles de protéines.
Ainsi, l’ensemble de ces travaux a permis d’identifier des protéines d’intérêt dont l’étude fonctionnelle est en cours. Actuellement, les projets concernent des protéines de fonction inconnue (DUF642), des protéines riches en Pro/Hyp, des lectines et des protéines liées au métabolisme des lipides.
Relations structure-fonction de protéines ayant des domaines d’interaction avec des constituants pariétaux
Contact : Elisabeth JAMET
Parallèlement aux approches systématiques d’identification des protéines pariétales, l’équipe tire profit de son expertise en protéomique et en biochimie pour élucider la structure et la fonction de certaines protéines pariétales d’intérêt. Ces dernières années, un intérêt particulier a été porté aux protéines de la famille DUF642 et à la protéine AGP31 (ArabinoGalactan Protein 31) d’Arabidopsis thaliana, codée par At1g28290, qui ont été identifiées en abondance dans les parois des hypocotyles étiolés, dont les cellules subissent une élongation rapide et importante.
La famille des protéines à domaine DUF642 (Domain of Unknown Function 642, PF04862) comporte 10 membres chez A. thaliana et est bien représentée dans tous les protéomes pariétaux (WallProtDB). Ces protéines sont constituées d’un peptide signal en N-terminal et de deux sous-domaines présentant des acides aminés conservés entre eux et structurés en feuillets béta. Certains membres de la famille possèdent une ancre GPI. Ces protéines ont été trouvées dans tous les génomes de spermatophytes analysés (Vasquez-Lobo, Roujol et al. 2012). Nous avons montré que l’une d’entre elle (At3g08030) interagit avec la cellulose in vitro (Vasquez-Lobo, Roujol et al. 2012).
AGP31 comporte un peptide signal en N-terminal, un domaine AGP, une région riche en His, un domaine central riche en Pro ainsi qu’un domaine C-terminal appelé PAC (PRP-AGP-containing Cys). Cette organisation multi-domaines est unique chez A. thaliana et fait d’AGP31 un excellent candidat pour une étude structurale et fonctionnelle.
La combinaison de différents approches de spectrométrie de masse et de biochimie ont permis de révéler la présence de résidus hydroxyproline (Hyp) et de O-glycanes riches en galactose et arabinose au niveau du domaine riche en Pro (Hijazi et al. 2012). AGP31 a été trouvée sous plusieurs glycoformes différant par le type et la taille des O-glycanes et/ou la longueur de la chaîne polypeptidique. En particulier, le domaine AGP peut porter des chaînes d’arabinogalaactanes (AG) et le domaine riche en Pro des O-glycanes dont la structure reste à élucider. Par ailleurs, des tests in vitro ont montré des interactions entre le domaine PAC d’AGP31 et des galactanes rencontrés sur le rhamnogalacturonane de type I (RG I) des pectines (Hijazi et al. 2014). Il a également été montré qu’AGP31 pouvait interagir avec elle-même, probablement via les O-glycanes de son domaine riche en Pro. Enfin, une remarquable affinité entre les O-glycanes du domaine riche en Pro et une lectine de cacahuète (PNA, PeaNut Agglutinin) a été mise en évidence, indiquant de possibles interactions avec des lectines pariétales.

L’ensemble des données obtenues a permis de proposer un modèle d’interactions non covalentes plaçant AGP31 au cœur d’un assemblage supramoléculaire complexe, suggérant un rôle structural pour cette protéine atypique. Ces résultats apportent des éléments nouveaux sur l’architecture de la paroi et met en lumière le rôle central des O-glycoprotéines dans son organisation (Hijazi et al. 2014).
Récepteurs lectine-kinases et Perception de modifications de l’intégrité pariétale
Contact : Hervé CANUT
Notre équipe s’intéresse à la communication entre les parois et le cytoplasme des cellules des plantes. Il s’agit de contribuer à la compréhension des fonctions des lectines chez la plante modèle Arabidopsis thaliana.
Les lectines sont des protéines présentes dans tous le règne vivant. Elles sont capables de se lier à des mono ou des oligosaccharides de manière réversible et de décoder l’information contenue dans ces structures complexes. Ces molécules, pour la plupart originaire des plantes, trouvent de multiples applications en sciences biologiques et en médecine. depuis l’application initiale pour le typage sanguin, des découvertes récentes ont permis de montrer leur rôle, souvent bénéfique, sur le développement des tumeurs et en thérapie anti-cancéreuse. De même, les lectines sont fondamentales pour la vie des plantes et jouent des rôles importants dans la communication inter-cellulaire, le développement et les stratégies naturelles de défense.
Les parois cellulaires végétales sont des structures complexes contenant de la cellulose, des hémicelluloses, des pectines et des protéines secrétées par les cellules, créant ainsi une structure rigide et continue dans les tissus des plantes. Ces parois sont dynamiques et sources de signaux chimiques ou mécaniques. Chez A. thaliana, les lectines sont présentes dans les parois, à la fois sous forme de protéines solubles faiblement liées aux parois (Lec), et sous forme chimérique localisées à la membrane plasmique : il s’agit alors de domaines extracelllulaires de lectine récepteurs kinases (LecRK) (Bellande et al. 2017).
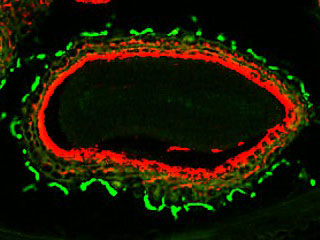
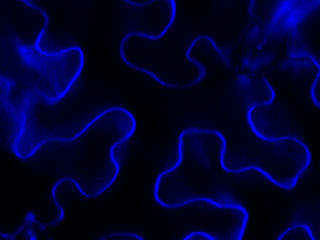
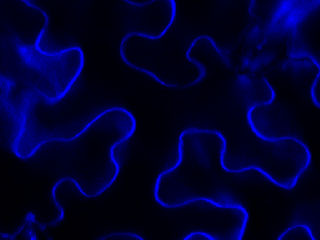
Nous avons montré que, chez A. thaliana, les contacts membrane plasmique-parois [1-flèche blanche] sont établis par des interactions protéine-protéine impliquant la reconnaissance du motif Arg-Gly-Asp (RGD). LecRK-I.9 (At5g60300) est l’un des candidats pour être un lien entre membrane plasmique et paroi (Gouget et al. 2006). Il appartient à la famille des lectines de type légumineuse [2]. Des résultats récents ont montré que la résistance à Pseudomonas syringae pv tomate (Pst), une bactérie pathogène et à Phytophthora infestans, est affectée chez un mutant lecrk-I.9 (Bouwmeester et al. 2011, Balagué et al. 2016).
Par ailleurs, notre analyse du protéome pariétal de cellules en élongation à montré la présence de plusieurs lectines (Irshad et al. 2008). des analyses phylogéniques ont montré que les protéines de type Lec et LecRK sont très proches et forment un cluster dans les arbres phylogéniques (Bellande et al. 2017). Enfin, des constructions « promoteur LecRK-I.9-GUS » ont montré que l’activité du promoteur de LecRK-I.9 est maximale dans les apex des racines principales et qu’elle est absente des primordia racinaires [3].

Le but de ce projet est de comprendre le rôle des protéines Lec/LecRK chez A. thaliana. Une combinaison d’approches de biochimie et de génomique fonctionnelle sera mise en œuvre pour identifier des ligands extracellulaires de ces protéines et élucider leur rôle. Nous supposons que les Lec/LecRK sont impliquées dans la détection de pathogènes, mais aussi dans l’assemblage et le remodelage des polysaccharides pariétaux. En particulier, les Lec/LecRK pourraient agir comme des senseurs de l’intégrité pariétale.
ANR : WALLARRAY (2009)
ANR : WALLARRAY2 (2011-2014)
Collaboration :
Francine GOVERS : Laboratory of Phytopathology, Wageningen University, Wageningen, Pays Bas.
Analyse fonctionnelle de CIII peroxydases pariétales au cours du développement chez Arabidopsis thaliana
Contact : Christophe DUNAND et Philippe RANOCHA (Equipe Peroxydases : évolution et expression)
Vincent Burlat (Equipe Protéines pariétales et Développement)
Contexte
Les 73 CIII Peroxydases d’Arabidopsis thaliana présentent des profils d’expression spécifiques et une prédiction de localisation subcellulaire pariétale ou vacuolaire en fonction de l’absence ou la présence d’un motif C-terminal d’adressage vacuolaire (Francoz et al. 2014). Des études récentes tendent à montrer que la localisation fine de peroxydases individuelles doit conduire à la rencontre spécifique « enzyme-substrat » permettant une action contrôlée au niveau de micro-domaines pariétaux. Cette action peut ensuite conceptuellement impliquer un rôle de relâchement pariétal ou de renforcement pariétal.

Dans le cadre d’un projet transversal impliquant les équipes « Peroxydases : évolution et expression » et « Protéines pariétales et Développement », nous étudions actuellement plus particulièrement le rôle de CIII peroxydases – principalement pariétales – au cours de deux processus développementaux conduisant à la formation de la graine et à sa germination. La sélection des candidats d’intérêt est basée sur une pre-sélection à travers l’analyse de données transcriptomiques et sur une sélection fine par hybridation d’ARN in situ systématique (Francoz et al. 2016). L’analyse fonctionnelle des candidats retenus présentant un profil d’expression spécifique est principalement menée par génétique inverse avec un intérêt important pour la localisation fine des protéines candidates et pour l’étude de microphénotypes.
Objectifs
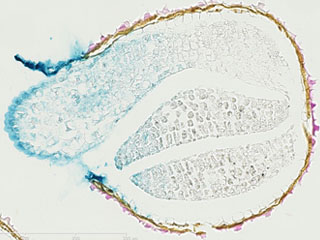
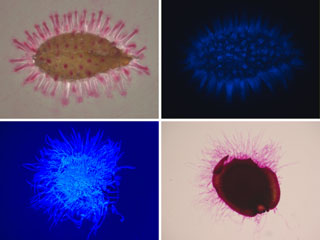
Contrôle de la dynamique pariétale des cellules sécrétrices de mucilage (MSC) de la graine
Au cours de l’embryogénèse les 5 couches cellulaires formant les téguments de la graine se spécialisent, la couche épidermique la plus externe présentant une dynamique pariétale remarquable. En effet, outre la paroi primaire principale, ces cellules synthétisent une paroi interne (columelle) en forme de volcan et produisent un abondant mucilage polysaccharidique comprimé entre la paroi primaire tangentielle externe et la columelle. Le rôle de ce mucilage ne se révèle que lors de l’imbibition de la graine plusieurs semaines à plusieurs années après la fin de la dessiccation de la graine. En effet, des renforcements ou relâchements localisés des différentes parois préalablement programmées lors de l’embryogénèse conduisent à une rupture polarisée de la paroi externe sous la pression du mucilage réhydraté. Une matrice pluristratifiée de mucilage entoure ainsi la graine favorisant le processus de germination. Nous recherchons actuellement la fonction, les modes de régulation et les cibles de plusieurs CIII Prx au cours de ce processus.
Contrôle de la rupture de l’endosperme micropylaire au cours de la germination
La germination est contrôlée par des facteurs externes comme la température, l’eau et la lumière et par l’équilibre hormonal. Deux étapes majeures de la germination sont la rupture des téguments de la graine puis la rupture de l’endosperme micropylaire permettant l’émergence de la radicule. Récemment, le rôle de messager secondaire des espèces réactives de l’oxygène (ROS) a été démontré au cours de ce processus (Lariguet et al. 2013). Nous avons montré que les ROS sont libérés avant la rupture de l’endosperme micropylaire, que la production de ROS est nécessaire à la germination, et que les CIII Prx pourraient être impliquées dans la régulation spatio-temporelle de la production de ROS au cours de ce processus. Nous étudions actuellement plus particulièrement plusieurs candidats CIII Prx pouvant être impliqués dans le contrôle de la rupture de l’endosperme micropylaire.
L’équipe Protéines pariétales et Développement a organisé :
- Les Journées du Réseau Français des Parois en 2008.

- La Journée annuelle du Protéome Vert en 2011.

- L’Ecole d’Été mixOmics dans le cadre du COST FA1306 The quest for tolerant varieties : phenotyping at plant and cellular level en 2016.
Elisabeth JAMET a été co-éditeur invité avec Els VAN DAMME, Nausicaa LANNOO et Cécile ALBENNE d’un e-book portant sur Plant Glycobiology – A sweet world of lectins, glycoproteins, glycolipids and glycans dans le cadre des Research Topics de Frontiers in Plant Science (2015) et co-éditeur invité d’un numéro spécial de Proteomes intitulé Plant Proteomics 2017 avec Véronique SANTONI. Elle a aussi été co-éditeur invité (i) avec Christophe DUNAND du Numéro Spécial de International Journal of Molecular Sciences (IJMS) intitulé Plant Cell wall Proteins and Development et (ii) avec Christophe DUNAND et Zoë POPPER d’un Special Topic de Frontiers in Plant Science portant sur Co-evolution of plant cell wall polymers.
E JAMET est actuellement co-éditeur invité avec Christophe DUNAND du Numéro Spécial de Cells intitulé Research on Plant cell Wall Biology, actuellement ouvert à la soumission de manuscrits.
Publications
2020
Bellande K, Lalo A, Ligat L, Roujol D, Jamet E, Canut H (2020) Recombinant N-glycosylation isoforms of Legume lectins: production and purification from Nicotiana benthamiana leaves following RuBisCO depletion. Plant Physiol Biochem 157: 441-452 (lire)
Cherkaoui M, Lollier V, Geairon A, Bouder A, Larré C, Rogniaux H, Jamet E, Guillon F, Francin-Allami M (2020) Cell wall proteome of wheat grain endosperm and outer layers at two key stages of early development. Int J Mol Sci 21: 239 (lire)
Duruflé H, Ranocha P, Balliau T, Zivy M, Albenne C, Burlat V, Déjean S, Jamet E, Dunand C (2020) An integrative study showing the adaptation to sub-optimal growth conditions of natural populations of Arabidopsis thaliana: a focus on cell wall changes. Cells 9: 2249 (lire)
Duruflé H, Selmani M, Ranocha P, Jamet E, Dunand C, Déjean S (2020) A powerful framework for an integrative study with heterogeneous omics data: from univariate statistics to multi-block analysis. Brief Bioinformatics bbaa166 (lire)
Roujol D, Hoffmann L, San Clemente H, Ritzenthaler C, Burlat V, Jamet E (2020) Plant cell wall proteomes : bioinformatics and cell biology tools to assess the bona fide cell wall localization of proteins. Methods Mol Biol 2149: 443-462 (lire)
Viudes S, Burlat V, Dunand C (2020) Seed mucilage evolution: diverse molecular mechanisms generate versatile ecological functions for particular environments. Plant Cell Environ (doi.org/10.1111/pce.13827) (lire)
2019
Berthold F, Roujol D, Hemmer C, Jamet E, Ritzenthaler C, Hoffmann L, Schmitt-Keichinger C (2019) Inside or outside? A new collection of Gateway vectors allowing plant protein subcellular localization or over-expression. Plasmid 105: 102436 (lire)
Francoz E, Ranocha P, Dunand C, Burlat V (2019). Medium-throughput RNA in situ hybridization of serial sections from paraffin-embedded tissue microarrays. Methods Mol Biol, 1933: 99-130 (lire)
Francoz E, Ranocha P, Le Ru A, Martinez Y, Fourquaux I, Jauneau A, Dunand C, Burlat (2019) Pectin demethylesterification generates platforms that anchor peroxidases to remodel plant cell wall domains. Dev Cell 48: 261-276 (lire)
Duruflé H, Albenne C, Jamet E, Dunand C (2019) Phenotyping and cell wall polysaccharide composition of five Arabidopsis ecotypes grown at optimal or sub-optimal temperatures. Data Brief 25: 104318 (lire)
Duruflé H, Ranocha P, Balliau T, Dunand C, Jamet E (2019) Transcriptomic and cell wall proteomic datasets of rosettes and floral stems from five Arabidopsis thaliana ecotypes grown at sub-optimal temperature. Data Brief 27: 104581 (lire)
Duruflé H, Selmani M, Ranocha P, Jamet E, Dunand C, Déjean S (2019) A powerful framework for an integrative study with heterogeneous omics data: from univariate statistics to multi-block analysis. bioRxiv (
2018
Calderan-Rodrigues MJ, Fonseca JG, San Clemente H, Labate CA, Jamet E (2018) Glycoside hydrolases in plant cell wall proteomes: predicting functions that could be relevant for improving biomass transformation processes. Advances in Biofuels and Bioenergy (M Nageshwara-Rao and J Soneji eds), InTechOpen access Publisher, Croatia, pp 165-182 (lire)
Cherkaoui M, Geairon A, Lollier V, San Clemente H, Larré C, Rogniaux H, Jamet E, Guillon F, Francin-Allami M (2018) Cell wall proteome investigation of bread wheat (Triticum aestivum) developing grain in endosperm and outer layers. Proteomics 18: 1800286 (lire)
Fonseca JG, Calderan-Rodrigues MJ, de Moraes FE, Cataldi TR, Jamet E, Labate CA (2018) Cell wall proteome of sugarcane young and mature leaves and stems. Proteomics 18: 1700129 (lire)
Jacq A, Burlat V, Jamet E (2018) Plant cell wall proteomics as a strategy to reveal candidate proteins involved in extracellular lipid metabolism. Curr Protein Pept Sci 19: 190-199 (lire)
Jamet E, Santoni V (2018) Editorial for Special Issue: 2017 Plant Proteomics. Proteomes 6: 28 (lire)
2017
Badruna L, Burlat V, Montanier C (2017) CBMs as probes to explore plant cell wall heterogeneity using immunocytochemistry. Methods Mol Biol 1588 : 181-197 (lire)
Balagué C, Gouget A, Bouchez O, Souriac C, Haget N, Boutet-Mercey S, Govers F, Roby D, Canut H (2016) The Arabidopsis thaliana lectin receptor kinase LecRK-I.9 is required for full resistance to Pseudomonas syringae and affects jasmonate signalling. Mol Plant Pathol 18 : 937-948 (lire)
Bellande K, Bono JJ, Savelli B, Jamet E, Canut H (2017) Plant lectins and lectin receptor-like kinases : How do they sense the outside ? Int J Mol Sci 18 : 1164 (lire)
Calderan-Rodrigues MJ, Fonseca JG, Labate CA, Jamet E (2017) Cell wall proteomics as a means to identify target genes to improve second generation biofuel production. In Frontiers in Bioenergy and Biofuels (E Jacob-Lopes and L Queiroz Zepka ed), InTechOpen access Publisher, Croatia, pp 5-24 (lire)
Canut H, Albenne C, Jamet E (2017) Isolation of the cell wall. Methods Mol Biol 1511 : 171-185 (lire)
Cosio C, Ranocha P, Francoz E, Burlat V, Zheng Y, Perry SE, Ripoll JJ, Yanofsky M, Dunand C (2017) The class III peroxidase PRX17 is a direct target of the MADS-box transcription factor AGAMOUS-LIKE15 (AGL15) and participates in lignified tissue formation. New Phytol 213 : 250-263 (lire)
Duruflé H, Hervé V, Balliau T, Zivy M, Dunand C, Jamet E (2017) Proline hydroxylation in cell wall proteins : is it yet possible to define rules ? Front Plant Sci 8: 1802 (lire)
Duruflé H, Hervé V, Ranocha P, Balliau T, Zivy M, Chourré J, San Clemente, H, Burlat V, Albenne C, Déjean S, Jamet E, Dunand C (2017) Cell wall modifications of two Arabidopsis thaliana ecotypes, Col and Sha, in response to sub-optimal growth conditions : an integrative study. Plant Sci 263 : 183-193 (lire)
Duruflé H, San Clemente H, Balliau T, Zivy M, Dunand C, Jamet E (2017) Cell wall proteome analysis of Arabidopsis thaliana mature stems. Proteomics 17, 8, 1600449 (lire)
Jacq A, Pernot C, Martinez Y, Domergue F, Payré B, Jamet E, Burlat V, Pacquit V (2017) The Arabidopsis Lipid Transfer Protein 2 (AtLTP2) is involved in cuticle-cell wall interface integrity and in etiolated hypocotyl permeability. Front Plant Sci 8 : 263 (lire)
Jamet E (2017) Plant cell wall proteomics : An assessment twenty years after launching. Bioinform. Proteomics Open Access J 1: 000107 (lire)
Regente M, Pinedo M, San Clemente H, Balliau T, Jamet E, de la Canal L (2017) Plant extracellular vesicles are incorporated by cells of a fungal pathogen and inhibit its growth. J Exp Bot 20: 5485-5496 (lire)
Sibout R, Proost S, Hansen B, Vaid N, Giorgi F, Ho-Yue-Kuang S, Legée F, Cezart L, Bouchabké-Coussa O, Soulhat C, Provart N, Pasha A, Le Bris P, Roujol D, Höfte H, Jamet E, Lapierre C, Persson S, Mutwil M (2017) Expression atlas and comparative co-expression network analyses reveal important genes involved in the formation of lignified cell wall in Brachypodium distachyon. New Phytol 215 : 1009-1025 (lire)
2016
Calderan-Rodrigues MJ, Jamet E, Douché T, Rodrigues Bonassi MB, Regiani Cataldi T, Fonseca Guimarães J, San Clemente H, Pont-Lezica R, Labate CA (2016) Cell wall proteome of sugarcane stems : comparison of a destructive and a non-destructive extraction method showed differences in glycoside hydrolases and peroxidases. BMC Plant Biol 16 : 14 (lire)
Canut H, Albenne C, Jamet E (2016) Post-translational modifications of plant cell wall proteins and peptides : A survey from a proteomics point of view. Biochim Biophys Acta 1864 : 983-990 (lire)
Francin-Allami M, Lollier V, Pavlovic M, San Clemente H, Rogniaux H, Jamet E, Guillon F, Larré C (2016) Understanding the remodelling of cell walls during Brachypodium distachyon grain development through a sub-cellular quantitative proteomic approach. Proteomes 4 : 21 (lire)
Francoz E, Ranocha P, Pernot C, Le Ru, A, Pacquit V, Dunand C, Burlat V (2016) Complementarity of medium-throughput in situ RNA hybridization and tissue-specific transcriptomics : case study of Arabidopsis seed development kinetics. Sci Rep 6:24644 (lire)
Hervé V, Duruflé H, San Clemente H, Albenne C, Balliau T, Zivy M, Dunand C, Jamet E (2016) An enlarged cell wall proteome of Arabidopsis thaliana rosettes. Proteomics 16 : 3183-3187 (lire)
Nguyen-Kim H, San Clemente H, Balliau T, Zivy M, Dunand C, Albenne C, Jamet E (2016) Arabidopsis thaliana root cell wall proteomics : increasing the proteome coverage using a combinatorial peptide ligand library and description of unexpected Hyp in peroxidase amino acid sequences. Proteomics 16 : 491-503 (lire)
2015
Dupoiron S, Zischek C, Ligat L, Carbonne J, Boulanger A, Dugé de Bernonville T, Lautier M, Rival P, Arlat M, Jamet E, Lauber E, Albenne C (2015) The N-glycan cluster from Xanthomonas campestris pv. campestris : a toolbox for sequential plant N-glycan processing. J Biol Chem 290 : 6022-6036 (lire)
Francin-Allami M, Merah K, Albenne C, Rogniaux H, Pavlovic M, Lollier V, Sibout R, Guillon F, Jamet E, Larré C (2015) Cell wall proteomics of Brachypodium distachyon grains : A focus on cell wall remodeling proteins. Proteomics 15 : 2296-2306 (lire)
Francoz E, Ranocha P, Burlat V, Dunand C (2015) Arabidopsis seed mucilage 1 secretory cells : Regulation and dynamics. Trends Plant Sci 20 : 515-524 (lire)
Francoz E, Ranocha P, Nguyen-Kim H, Jamet E, Burlat V, Dunand C (2015) Roles of cell wall peroxidases in plant development. Phytochemistry 112 : 15-21 (lire)
Pont-Lezica RF (2015) Localizing proteins by tissue printing. Methods Mol Biol 1312 : 93-104 (lire)
San Clemente H, Jamet E (2015) WallProtDB, a database resource for plant cell wall proteomics. Plant Methods 11 : 2 (lire)
Sénéchal F, L’Enfant M, Domon JM, Rosiau E, Crépeau MJ, Surcouf O, Esquivel-Rodriguez J, Marcelo P, Mareck A, Guérineau F, Hyung-Rae K, Mravec J, Bonnin E, Jamet E, Kihara D, Lerouge P, Ralet MC, Pelloux J, Rayon C (2015) Tuning of pectin methylesterification : PECTIN METHYLESTERASE INHIBITOR 7 modulates the processive activity of co-expressed PECTIN MEHYLESTERASE 3 in a pH-dependent manner. J Biol Chem 290 : 23320-23335 (lire)
2014
Albenne C, Canut H, Hoffmann L, Jamet E (2014) Plant cell wall proteins : a large body of data, but what about runaways ? Proteomes 2 : 224-242 (lire)
Calderan-Rodrigues MJ, Jamet E, Calderan Rodrigues Bonassi MB, Guidetti-Gonzalez S, Carmanhanis Begossi A, Vaz Setem L, Franceschini LM, Guimarães Fonseca J, Labate CA (2014) Cell wall proteomics of sugarcane cell suspension cultures. Proteomics 14 : 738-749 (lire)
Dugé de Bernonville T, Albenne C, Arlat M, Hoffmann L, Lauber E, Jamet E (2014) Xylem sap proteomics. Methods Mol Biol 1072 : 391-405 (lire)
Hijazi M, Roujol D, Nguyen-Kim H, del Rocio Cisneros Castillo L, Saland E, Jamet E, Albenne C (2014) Arabinogalactan protein 31 (AGP31), a putative network-forming protein in Arabidopsis thaliana cell walls ? Ann Bot 114 : 1087-1097 (lire)
Hijazi M, Velasquez MS, Jamet E, Estevez JM, Albenne C (2014) An update on post-translational modifications of hydroxyproline-rich glycoproteins : Towards a model highlighting their contribution to plant cell wall architecture. Front Plant Sci 5 : 395 (lire)
Lannoo N, Van Damme EJM, Albenne C, Jamet E (2014) Plant Glycobiology – a diverse world of lectins, glycoproteins, glycolipids and glycans. Front Plant Sci 5 : 604 (lire). Accès e-book.
Ranocha P, Francoz E, Burlat V, Dunand C (2014) Expression of PRX36, PMEI6 and SBT1.7 is controlled by complex transcription factor regulatory networks for proper seed coat mucilage extrusion. Plant Signal Behav 9 : e977734 (lire)
2013
Albenne C, Canut H, Jamet E (2013) Plant cell wall proteomics : the leadership of Arabidopsis thaliana. Front Plant Sci 4 : 111 (lire)
Douché T, San Clemente H, Burlat V, Roujol D, Valot B, Zivy M, Pont-Lezica R, Jamet E (2013) Brachypodium distachyon as a model plant towards improved biofuel crops : Search for secreted proteins involved in biogenesis and disassembly of cell wall polymers. Proteomics 13 : 2438-2454 (lire)
Mallèvre F, Roget A, Kervella Y, Ropartz D, Ralet M-C, Canut H, Minon T, Livache T (2013) Microwave heating for the rapid generation of glycosylhydrazides. Bioconjugate Chem 24 : 1264-1269 (lire)
Simkin AJ, Miettinen K, Claudel P, Burlat V, Guirimand G, Courdavault V, Papon N, Meyer S, Godet S, St-Pierre B, Giglioli-Guivarc’h N, Fischer MJ, Memelink J, Clastre M (2013) Characterization of the plastidial geraniol synthase from Madagascar periwinkle which initiates the monoterpenoid branch of the alkaloid pathway in internal phloem associated parenchyma. Phytochemistry 85:36-43
2012
Geu-Flores F, Sherden NH, Courdavault V, Burlat V, Glenn WS, Wu C, Nims E, Cui Y, O’Connor SE (2012) An alternative route to cyclic terpenes by reductive cyclization in iridoid biosynthesis. Nature 492:138-142
Guirimand G, Guihur A, Phillips M, Oudin A, Glévarec G, Mahroug S, Melin C, Papon N, Clastre M, Giglioli-Guivarc’h N, St-Pierre B, Rodríguez-Concepción M, Burlat V, Courdavault V (2012) Triple subcellular targeting of isopentenyl diphosphate isomerases encoded by a single gene. Plant Signal Behav 7 : 1495-1497
Guirimand G, Guihur A, Phillips MA, Oudin A, Glévarec G, Melin C, Papon N, Clastre M, St-Pierre B, Rodríguez-Concepción M, Burlat V, Courdavault V (2012) A single gene encodes isopentenyl diphosphate isomerase isoforms targeted to plastids, mitochondria and peroxisomes in Catharanthus roseus. Plant Mol Biol 79 : 443-459
Guirimand G, Simkin AJ, Papon N, Besseau S, Burlat V, St-Pierre B, Giglioli-Guivarc’h N, Clastre M, Courdavault V (2012) Cycloheximide as a tool to investigate protein import in peroxisomes : a case study of the subcellular localization of isoprenoid biosynthetic enzymes. J Plant Physiol 169 : 825-829
Hijazi M, Durand J, Pichereaux C, Pont F, Jamet E, Albenne C (2012) Characterization of the ArabinoGalactan Protein 31 (AGP31) of Arabidopsis thaliana : new advances on the Hyp-O-glycosylation of the Pro-rich domain. J Biol Chem 287 : 9623-9632 (lire)
Vázquez-Lobo A, Roujol D, Zuñiga-Sánchez E, Albenne C, Piñero D, Gamboa de Buen A, Jamet E (2012) The highly conserved spermatophyte cell wall DUF642 protein family : phylogeny and first evidence of interaction with cell wall polysaccharides in vitro. Mol Phylogenet Evol 63 : 510-520 (lire)
2011
Bouwmeester K, de Sain M, Weide R, Gouget A, Klamer S, Canut H, Govers F (2011) The lectin receptor kinase LecRK-I.9 is a novel Phytophthora resistance component and a host target for a RXLR effector. PLoS Pathog 7 : e1001327 (lire)
Guirimand G, Guihur A, Ginis O, Poutrain P, Héricourt F, Oudin A, Lanoue A, St-Pierre B, Burlat V, Courdavault V (2011) The subcellular organization of strictosidine biosynthesis in Catharanthus roseus epidermis highlights several trans-tonoplast translocations of intermediate metabolites. FEBS J 278 : 749-63
Guirimand G, Guihur A, Poutrain P, Héricourt F, Mahroug S, St-Pierre B, Burlat V, Courdavault V (2011) Spatial organization of the vindoline biosynthetic pathway in Catharanthus roseus. J Plant Physiol 6 : 549-557
Guénin S, Mareck A, Rayon C, Lamour R, Assoumou, Ndong Y, Domon JM, Sénéchal F, Fourneta F, Jamet E , Canut H, Percoco G, Mouille G, Rolland A, Rustérucci C, Guérineau F, Van Wuytswinkel O, Gillet F, Driouich A, Lerouge P, Gutierrez L, Pelloux J (2011) Identification of PME3 as a basic pectin methylesterase isoform involved in adventitious rooting in Arabidopsis. New Phytol 192 : 114-126 (lire)
Jamet E (2011). Obituary : Rafael Pont-Lezica. Front Plant Sci 2:86
Ligat L, Lauber E, Albenne C, San Clemente H, Valot B, Zivy M, Pont-Lezica R, Arlat M, Jamet E (2011) Analysis of the xylem sap proteome of Brassica oleracea reveals a high content in secreted proteins. Proteomics 11 : 1798-1813 (lire)
Poutrain P, Guirimand G, Mahroug S, Burlat V, Melin C, Ginis O, Oudin A, Giglioli-Guivarc’h N, Pichon O, Courdavault V (2011) Molecular cloning and characterisation of two calmodulin isoforms of the Madagascar periwinkle Catharanthus roseus. Plant Biol 13 : 36-41
Zhang Y, Giboulot A, Zivy M, Valot B, Jamet E, Albenne C (2011) Combining various strategies to increase the coverage of the plant cell wall glycoproteome. Phytochemistry 72 : 1109-1123 (lire)
2010
Guirimand G, Courdavault V, Lanoue A, Mahroug S, Guihur A, Blanc N, Giglioli-Guivarc’h N, St-Pierre B, Burlat V (2010) Strictosidine activation in Apocynaceae : towards a « nuclear time bomb » ? BMC Plant Biol 10 : 20
Guirimand G, Courdavault V, St-Pierre B, Burlat V (2010) Biosynthesis and regulation of alkaloids. In E Pua, M Davey, eds, Plant developmental biology – Biotechnological perspectives, vol 2. Springer-Verlag, Berlin Heidelberg, pp 139-160
Jamet E, Pont-Lezica R (2010) The puzzle of protein location in plant proteomics. In G Rancourt, ed, Proteomics : Methods, Applications and Limitations. Nova Science Publishers, Inc., Hauppauge, NY, USA. (lire)
Lanoue A, Burlat V, Henkes GJ, Koch I, Schurr U, Rose USR (2010) De novo biosynthesis of defense root exudates in response to Fusarium attack in barley. New Phytol 185 : 577-588
Lanoue A, Burlat V, Schurr U, Rose US (2010) Induced root-secreted phenolic compounds as a belowground plant defense. Plant Signal Behav 5 : 1037-1038
Pont-Lezica R, Minic Z, San Clemente H, Roujol D, Jamet E (2010) Plant cell wall functional genomics : Novelties from proteomics. In M Osborne, ed, Advances in Genetics Research, vol 1. Nova Science Publishers, Inc, Hauppauge, NY, USA, pp 265-281. (lire)
2009
Albenne C, Canut H, Boudart G, Zhang Y, San Clemente H, Pont-Lezica R, Jamet E (2009) Plant cell wall proteomics : mass spectrometry data, a trove for research on protein structure/function relationships. Mol Plant 2 : 977-989. (lire)
Courdavault V, Burlat V, St-Pierre B, Giglioli-Guivarc’h N (2009) Proteins prenylated by type I protein geranylgeranyltransferase act positively on the jasmonate signalling pathway triggering the biosynthesis of monoterpene indole alkaloids in Catharanthus roseus. Plant Cell Rep 28 : 83-93
Guirimand G, Burlat V, Oudin A, Lanoue A, St-Pierre B, Courdavault V (2009) Optimization of the transient transformation of Catharanthus roseus cells by particle bombardment and its application to the subcellular localization of hydroxymethylbutenyl 4-diphosphate synthase and geraniol 10-hydroxylase. Plant Cell Rep 28 : 1215-1234
Jamet E, Roujol D, San Clemente H, Irshad M, Soubigou-Taconnat L, Renou J, Pont-Lezica R (2009) Cell wall biogenesis of Arabidopsis thaliana elongating cells : transcriptomics complements proteomics. BMC Genomics 10 : 505 (lire)
Minic Z, Jamet E, San Clemente H, Pelletier S, Renou J, Rihouey C, Okinyo D, Proux C, Lerouge P, Jouanin L (2009) Transcriptomic analysis of Arabidopsis developing stems : a close-up on cell wall genes. BMC Plant Biol 9 : 17 (lire)
Pont-Lezica RF (2009) Localizing proteins by tissue printing. Methods Mol Biol 536 : 75-88 (lire)
San Clemente H, Pont-Lezica R, Jamet E (2009) Bioinformatics as a tool for assessing the quality of sub-cellular proteomic strategies and inferring functions of proteins : plant cell wall proteomics as a test case. Bioinform Biol Insights 3 : 15-28 (lire)
2008
Irshad M, Canut H, Borderies G, Pont-Lezica R, Jamet E (2008) A new picture of cell wall protein dynamics in elongating cells of Arabidopsis thaliana : confirmed actors and newcomers. BMC Plant Biol 8 : 94 (lire)
Jamet E, Albenne C, Boudart G, Irshad M, Canut H, Pont-Lezica R (2008) Recent advances in plant cell wall proteomics. Proteomics 8 : 893-908 (lire)
Jamet E, Boudart G, Borderies G, Charmont S, Lafitte C, Rossignol M, Canut H, Pont-Lezica R (2008) Isolation of plant cell wall proteins. Methods Mol Biol 425 : 187-201 (lire)
Jamet E, Canut H, Albenne C, Boudart G, Pont-Lezica R (2008) Cell wall. In G Agrawal, R Rakwal, eds, Plant Proteomics : Technologies, Strategies, and Applications. Wiley, USA, pp 293-307. (lire)
Jouili H, Bouazizi H, Rossignol M, Borderies G, Jamet E, El Ferjani E (2008) Partial purification and characterization of a copper-induced anionic peroxidase of sunflower roots. Plant Physiol Biochem 46 : 760-767 (lire)
2007
Boudart G, Minic Z, Albenne C, Canut H, Jamet E, Pont-Lezica R (2007) Cell wall proteome. In S Samaj, J Thelen, eds, Plant proteomics. Springer, Berlin, pp 169-185 (lire)
Minic Z, Jamet E, Négroni L, der Garabedian AP, Zivy M, Jouanin L (2007) A sub-proteome of Arabidopsis thaliana mature stems trapped on Concanavalin A is enriched in cell wall glycoside hydrolases. J Exp Bot 58 : 2503-2512 (lire)
2006
Feiz L, Irshad M, Pont-Lezica R, Canut H, Jamet E (2006) Evaluation of cell wall preparations for proteomics : a new procedure for purifying cell walls from Arabidopsis hypocotyls. Plant Methods 2 : 10 (lire)
Gouget A, Senchou V, Govers F, Sanson A, Barre A, Rougé P, Pont-Lezica R, Canut H (2006) Lectin receptor kinases participate in protein-protein interactions to mediate plasma membrane-cell wall adhesions in Arabidopsis. Plant Physiol 140 : 81-90 (lire)
Jamet E, Canut H, Boudart G, Pont-Lezica R (2006) Cell wall proteins : a new insight through proteomics. Trends Plant Sci 11 : 33-39 (lire)
2005
Aubourg S, Brunaud V, Bruyere C, Cock M, Cooke R, Cottet A, Couloux A, Dehais P, Deleage G, Duclert A, Echeverria M, Eschbach A, Falconet D, Filippi G, Gaspin C, Geourjon C, Grienenberger J-M, Houlné G, Jamet E, Lechauve F, Leleu O, Leroy P, Mache R, Meyer C, Nedjari H, Negrutiu I, Orsini V, Peyretaillade E, Pommier C, Raes J, Risler J-L, Rivière S, Rombault S, Rouzé P, Schneider M, Schwob P, Small I, Soumayet-Kampetanga G, Stankovski D, Toffano C, Tognolli M, Caboche M, Lecharny A (2005) GENEFARM, structural and functional annotation of Arabidopsis gene and protein families by a network of experts. Nucleic Acids Res 33 : D641-D646.
Boudart G, Jamet E, Rossignol M, Lafitte C, Borderies G, Jauneau A, Esquerré-Tugayé M, Pont-Lezica R (2005) Cell wall proteins in apoplastic fluids of Arabidopsis thaliana rosettes : identification by mass spectrometry and bioinformatics. Proteomics 5 : 212-221. (lire)
Charmont S, Jamet E, Pont-Lezica R, Canut H (2005) Proteomic analysis of secreted proteins from Arabidopsis thaliana seedlings : improved recovery following removal of phenolic compounds. Phytochemistry 66 : 453-461. (lire)
Masclaux F, Galaud J, Pont-Lezica R (2005) The riddle of the plant vacuolar sorting receptors. Protoplasma 226 : 103-108. (lire)
Masclaux F, Pont-Lezica R, Galaud J (2005) Relationship between allelic state of T-DNA and DNA methylation of chromosomal integration region in transformed Arabidopsis thaliana plants. Plant Mol Biol 58 : 295-303. (lire)
Schantz M, Jamet E, Guitton A, Schantz R, Houlné G (2005) Functional analysis of the bell pepper knolle gene (cakn) promoter region in tobacco plants and in synchronized BY2 cells. Plant Sci 169 : 155-163.
2004
Guzzardi P, Genot G, Jamet E (2004) The Nicotiana sylvestris extensin gene, Ext 1.2A, is expressed in the root transition zone and upon wounding. Biochim Biophys Acta 1680 : 83-92. (lire)
Jamet E (2004) Bioinformatics as a critical prerequisite to transcriptome and proteome studies. J Exp Bot 55 : 1977-1979. (lire)
Masclaux F, Charpenteau M, Takahashi T, Pont-Lezica R, Galaud J (2004) Gene silencing using a heat-inducible RNAi system in Arabidopsis. Biochem Biophys Res Commun 321 : 364-369. (lire)
Senchou V, Weide R, Carrasco A, Bouyssou H, Pont-Lezica R, Govers F, Canut H (2004) High affinity recognition of a Phytophthora protein by Arabidopsis via an RGD motif. Cell Mol Life Sci 61 : 502-509. (lire)
2003
Borderies G, Jamet E, Lafitte C, Rossignol M, Jauneau A, Boudart G, Monsarrat B, Esquerré-Tugayé M, Boudet A, Pont-Lezica R (2003) Proteomics of loosely bound cell wall proteins of Arabidopsis thaliana cell suspension cultures : a critical analysis. Electrophoresis 24 : 3421-3432. (lire)
Laval V, Masclaux F, Serin A, Carrière M, Roldan C, Devic M, Pont-Lezica R, Galaud J (2003) Seed germination is blocked in Arabidopsis putative vacuolar sorting receptor (atbp80) antisense transformants. J Exp Bot 54 : 213-221
Salva I, Jamet E (2003) The tobacco Ext 1.4 extensin gene family is not regulated in cells proliferating under hormone control Plant Physiol Biochem 41 : 363-367 (lire)
Thèses
Adélaïde Jacq (2013-2016) Université Paul Sabatier, Toulouse 3. Caractérisation fonctionnelle d’AtLTP2, une protéine de transfert de lipides impliquée dans le contrôle de l’intégrité de la cuticule chez Arabidopsis thaliana. (lire)
Edith Francoz (2012-2015) Université Paul Sabatier, Toulouse 3. Rôle de la famille multigénique des peroxydases pariétales de classe III dans le développement et la dynamique des parois cellulaires végétales. (lire)
Prix de thèse Henri Gaussen de l’Académie des Sciences Inscriptions et Belles lettres de Toulouse (2016) (lire)
Huan Nguyen-Kim (2011-2015) Université Paul Sabatier, Toulouse 3. Recherche de la fonction de protéines riches en hydroxyproline dans les parois végétales. (lire)
May Hijazi (2011) Université Paul Sabatier, Toulouse 3. Caractérisation structurale et fonctionnelle d’AGP31, une glycoprotéine atypique de la paroi chez Arabidopsis thaliana. (lire)
Muhammad Irshad (2008) Université Paul Sabatier, Toulouse 3. Dynamique des protéines pariétales au cours de l’élongation cellulaire dans des hypocotyles étiolés d’Arabidopsis thaliana : approches protéomique et transcriptomique. (lire)
Anne Gouget (2006) Université Paul Sabatier, Toulouse 3. Etude fonctionnelle d’un récepteur lectine kinase (LecRK79), potentiel partenaire dans les contacts paroi-plasmalemme chez Arabidopsis thaliana.
lrsv.gestion@univ-tlse3.fr
Phone
Standard : 05.34.32.38.01
Fax : 05.34.32.38.02
Find us
24, chemin de Borde-Rouge.
31320, Auzeville-Tolosane. FRANCE









